张晓轩、倪宏波教授团队在动物肠道微生物研究领域取得新进展
近日,青岛农业大学动物医学院张晓轩、倪宏波教授团队在微生物学综合期刊《Microbiology Spectrum》(IF 9.043,1区top)发表2篇研究型论文“A Catalog of over 5,000 Metagenome-Assembled Microbial Genomes from the Caprinae Gut Microbiota”和“Description of gut mycobiota composition and diversity of Caprinae animals(Accepted)”,通过宏基因组学测序和ITS扩增子测序技术深度挖掘了羊科动物肠道细菌和真菌组成,极大扩展了羊肠道微生物基因库。青岛农业大学为论文第一单位和主要通讯单位。

张晓轩、倪宏波课题组使用超深(每个样本>100Gbp)宏基因组测序技术,对来自中国六个省的30只绵羊和山羊的肠道微生物群进行研究,构建了包含5046个宏基因组组装基因组(MAG)的山羊肠道微生物物种清单。其中3263(98.7%)个MAG被分配到细菌门,包括厚壁菌门、拟杆菌门、变形杆菌门、疣状杆菌门、放线菌门、黏胶球形菌门、螺旋菌门和其他10个门,挖掘出2530个未被发现的潜在新菌。这些基因组极大地扩展了羊科动物肠道中现有微生物的基因组库。
研究大幅扩展了羊的肠道微生物群特征,丰富了对羊肠线菌群的分类、功能和代谢多样性的理解,并为未来的微生物起源和衍化等相关基础研究、动物生产和动物健康研究提供了基础。同时提出了反刍动物消化道菌群可能作为多种细菌的进化库的假设,为反刍动物消化道微生物相关研究提供了新的方向。
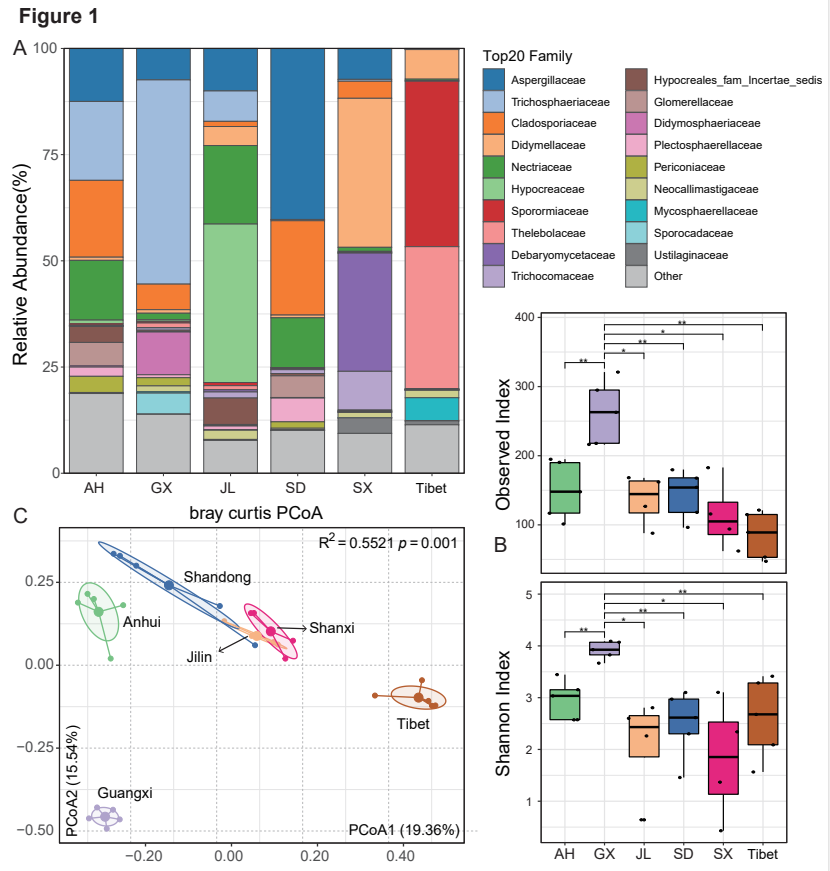
通过真菌ITS-2扩增子测序技术对不同地理位置的羊肠道真菌菌群的组成和多样性进行了综合表征,首次预测孢子菌科和铁杉科可能在羊高原适应性中起到重要作用,并首次揭示了羊的肠道细菌和真菌的互作网络。研究深入阐明了繁杂的反刍动物的肠道菌群多样性,发现了众多新真菌(未注释到种),为后续反刍动物消化道微生物的资源挖掘与功能研究提供了新的方向。
该研究得到青岛农业大学高层次引进人才项目等资金支持。张晓轩、倪宏波教授课题组长期致力于动物肠道微生物资源挖掘与利用、动物重要疫病防控相关研究工作,在相关领域取得一系列的科研成果,主持国家自然科学基金面上项目、国家自然科学基金青年项目、国家重点研发计划项目等多项项目。
论文链接:https://journals.asm.org/doi/pdf/10.1128/spectrum.02211-22

 地址:山东省 青岛市 城阳区 长城路700号
地址:山东省 青岛市 城阳区 长城路700号
 电话:0532-58957222
电话:0532-58957222
 邮编:266109
邮编:266109
 邮箱:xwzx@qau.edu.cn
邮箱:xwzx@qau.edu.cn














.jpg)
.jpg)
.jpg)
.jpg)
.jpg)
.jpg)
.png)
.jpg)

.png)

